Этиопатогенетическая архитектура разработки лекарственных средств (на примере этиопатогенеза подагры)
https://doi.org/10.30895/1991-2919-2025-724
Резюме
ВВЕДЕНИЕ. Системное понимание патофизиологических и патобиохимических путей заболеваний и патологических состояний является насущной потребностью как для врачей при постановке диагноза и лечении конкретного пациента, так и для разработчиков лекарственных средств (ЛС). Накопленные знания в медицинской и в смежных сферах, быстрое развитие цифровых инструментов делают возможным на качественно новом уровне смоделировать систему реактивности организма в норме и патологии. Это приведет к созданию цифровой архитектуры состояний организма с взаимосвязанными звеньями патогенеза, которые и будут находиться в фокусе внимания исследователей от дизайна ранней разработки ЛС до клинических исследований.
ЦЕЛЬ. Ревизия имеющихся подходов и построение на их основе этиопатогенетической архитектуры состояний и заболеваний как фундаментальной основы целенаправленной разработки лекарственных средств.
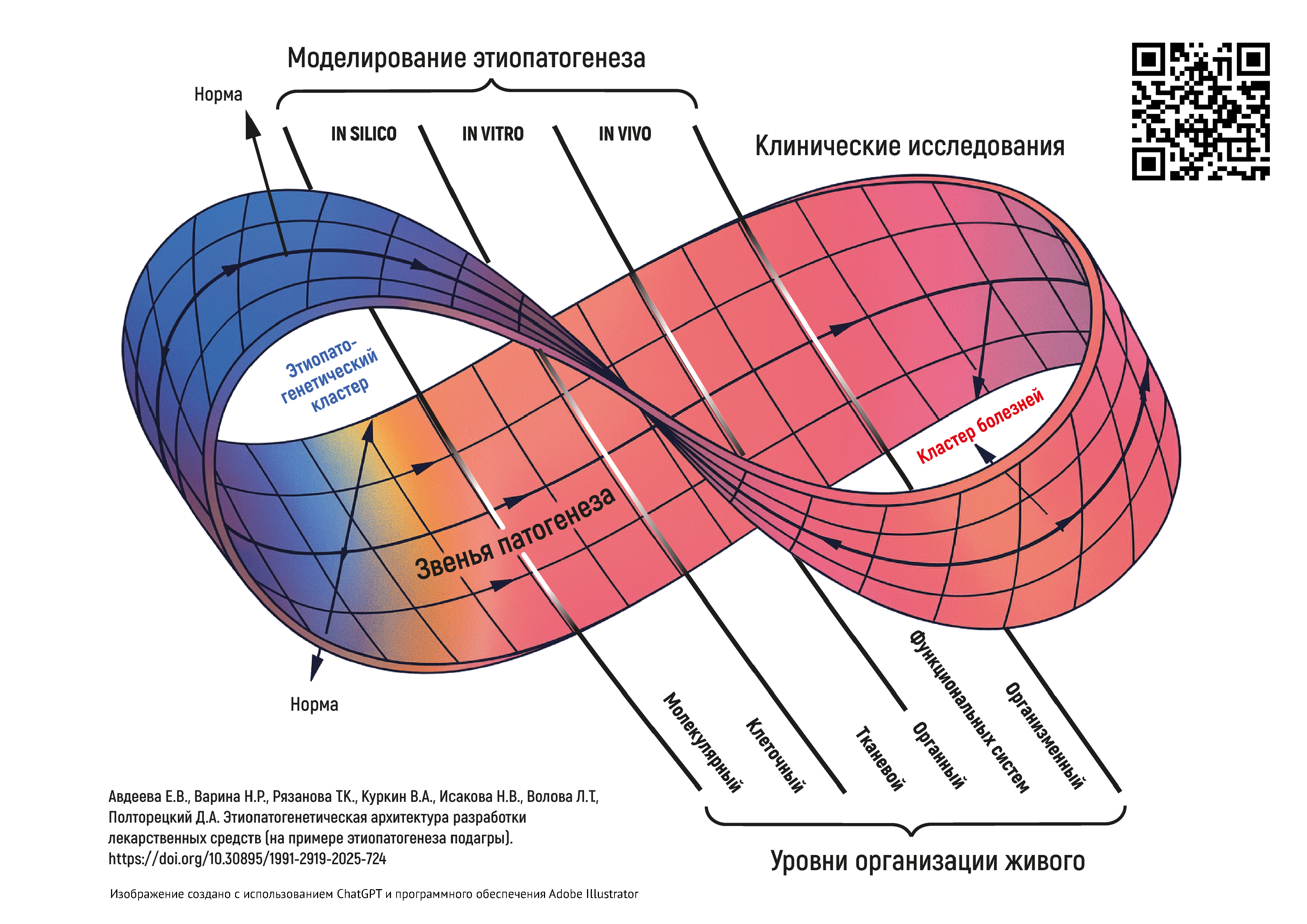
ОБСУЖДЕНИЕ. В исследовании были показаны необходимость и возможность (на примере подагры) построения объемной этиопатогенетической архитектуры состояний и заболеваний организма, основанной на иерархических связях патологических процессов на разных уровнях организации живого. Обоснованы основные векторы ее использования: для медицинских целей — основанная на данных диагностика состояний и заболеваний индивида, персонификация фармакотерапии; для фармацевтических целей — основа для исследования фармакодинамики ЛС начиная со скрининга веществ-кандидатов, использования методологических возможностей таргетных и мультитаргетных подходов в разработке ЛС. На примере архитектуры этиопатогенеза подагры обсуждена логика в разработке дизайна исследования ЛС.
ВЫВОДЫ. Предложена методология построения этиопатогенетической архитектуры как отражение причинно-следственных связей с разным уровнем значимости в формировании патологических состояний и заболеваний организма. Этиопатогенетический подход должен стать связующей основой между всеми этапами создания и применения новых ЛС, а также при исследовании возможности расширения показаний к применению уже использующихся ЛС. Появляются новые возможности и для разработки этиопатогенетических моделей разных уровней сложности: от drug-дизайна на молекулярном уровне до моделирования патофизиологических процессов на организменном уровне.
Ключевые слова
Об авторах
Е. В. АвдееваРоссия
Авдеева Елена Владимировна, д-р фарм. наук, профессор
ул. Чапаевская, д. 89, г. Самара, 443079
Н. Р. Варина
Россия
Варина Наталья Рашидовна, канд. фарм. наук
ул. Чапаевская, д. 89, г. Самара, 443079
Т. К. Рязанова
Россия
Рязанова Татьяна Константиновна, д-р фарм. наук, доцент
ул. Чапаевская, д. 89, г. Самара, 443079
В. А. Куркин
Россия
Куркин Владимир Александрович, д-р фарм. наук, профессор
ул. Чапаевская, д. 89, г. Самара, 443079
Н. В. Исакова
Россия
Исакова Наталья Викторовна, канд. мед. наук
ул. Чапаевская, д. 89, г. Самара, 443079
Л. Т. Волова
Россия
Волова Лариса Теодоровна, д-р мед. наук, профессор
ул. Чапаевская, д. 89, г. Самара, 443079
Д. А. Полторецкий
Россия
Полторецкий Денис Александрович
ул. Чапаевская, д. 89, г. Самара, 443079
Список литературы
1. Дедов ИИ. Персонализированная медицина. Вестник Российской академии медицинских наук. 2019;74(1):61–70. https://doi.org/10.15690/vramn1108
2. Долгополов ИС, Рыков МЮ. Эволюция персонализированной медицины: обзор литературы. Исследования и практика в медицине. 2022;9(3):117–28. https://doi.org/10.17709/2410-1893-2022-9-3-9
3. Хохлов АЛ, Сычев ДА. Концепция пациентоориентированности в медицине и фармации. Пациентоориентированная медицина и фармация. 2023;1(1):1–4. https://doi.org/10.37489/2949-1924-0001
4. Щербо СН., Щербо ДС. Лабораторная диагностика как основа медицины 5П. Вестник РГМУ. 2019;(1):5–14. https://doi.org/10.24075/brsmu.2018.095
5. Trocin C, Mikalef P, Papamitsiou Z, Conboy K. Responsible AI for digital health: A synthesis and a research agenda. Inf Syst Front. 2023;25:2139–57. https://doi.org/10.1007/s10796-021-10146-4
6. Германов НС. Концепция ответственного искусственного интеллекта — будущее искусственного интеллекта в медицине. Digital Diagnostics. 2023;4(1S):27–9. https://doi.org/10.17816/DD430334
7. Колсанов АВ, Седашкина ОА, Порецкова ГЮ, Плахотникова СП, Постников МА, Бесчастная ЕО. Клинико-параклинические и молекулярно-генетические показатели детей с хронической болезнью почек, подлежащие диспансерному наблюдению в Самарской области. Свидетельство о регистрации базы данных Российской Федерации № 2024622010; 2024. EDN: QTUISW
8. Авдеева ЕВ, Воронин АС, Ерхов ВЮ. Программа для изучения медицинских дисциплин по принципу мультидисциплинарного нозологического подхода «3D структура знания». Свидетельство о регистрации программы для ЭВМ Российской Федерации № 2021619194; 2021. EDN: PQKMJX
9. Пахомов ВА, Соболевская ПА, Кузнецов АВ, Чурилов ЛП, Норматов МГ. Программа для анализа белков на содержание перекрывающихся аминокислотных последовательностей с белками-аллергенами человека (Allergentaj). Свидетельство о регистрации программы для ЭВМ Российской Федерации № 2023685928; 2023.
10. Малышев ИЮ, Пихлак АЭ, Буданова ОП. Молекулярные и клеточные механизмы воспаления при подагре. Патогенез. 2019;17(4):4–13. https://doi.org/10.25557/2310-0435.2019.04.4-13
11. Гаврилова НЮ, Бреговская АВ, Сопрун ЛА, Лукашенко МВ, Чурилов ЛП, Федоткина ТВ, Пчелин ИЮ. Способ диагностики ранних неврологических нарушений у пациентов с сахарным диабетом 1 типа. Патент Российской Федерации № 2808480; 2023. EDN: GEOKMI
12. Павлова ВЮ, Денисенко ВЕ, Чеснокова ЛД, Анешина ИИ. Диагностические возможности исследования мочи. Фундаментальная и клиническая медицина. 2022;7(4):122–35. https://doi.org/10.23946/2500-0764-2022-7-4-122-135
13. Kang HS, Lee N-E, Yoo DM, Han KM, Hong JY, Choi HG, et al. An elevated likelihood of stroke, ischemic heart disease, or heart failure in individuals with gout: A longitudinal follow-up study utilizing the National Health Information data-base in Korea. Front Endocrinol (Lausanne). 2023;14:1195888. https://doi.org/10.3389/fendo.2023.1195888
14. Качковский МА, Федорина ТА, Рагозина ЕЮ, Чекулдаева ЛЕ, Глубоков ДГ, Суворов АЕ. Оценка влияния интенсивности системной воспалительной реакции на размер некроза миокарда по данным морфометрического исследования. Морфологические ведомости. 2013;(3):53–6. EDN: SDFRPV
15. Яшин СС, Киреева АО, Сухачев ПА. Стресс-индуцированная кардиомиопатия (синдром Такоцубо). Инновационная медицина Кубани. 2023;8(1):111–5. https://doi.org/10.35401/2541-9897-2023-26-1-111-115
16. Дадали ВА. Роль кобальта, меди, молибдена, селена, цинка, марганца в обеспечении реакций гомеостатического регулирования. Терапевт. 2022;(11):36–9. https://doi.org/10.33920/MED-12-2211-04
17. Гонсалвеш К, Маседо Х, Фернандеш Л, Чурилов Л, Карвалью Ж. Пробиотики при ревматических заболеваниях: систематический обзор. Российские биомедицинские исследования. 2023;8(2):92–111. https://doi.org/10.56871/RBR.2023.39.42.011
18. Елисеев МС. Классификационные критерии подагры (рекомендации ACR/EULAR). Научно-практическая ревматология. 2015;53(6):581–5. https://doi.org/10.14412/1995-4484-2015-581-585
19. Либов ИА, Моисеева ЮН, Комарова АГ. Гиперурикемия как фактор риска сердечно-сосудистых осложнений у пациентов с артериальной гипертонией. Российский кардиологический журнал. 2022;27(9):5194. https://doi.org/10.15829/1560-4071-2022-5194
20. Ivanov MF, Balmasova IP, Zhestkov AV, Rogozina LA. The role of innate and adaptive immune response lymphocytes in pathogenesis of hemorrhagic fever with renal syndrome. ACTA Sci Med Sci. 2019;3(8):120. https://doi.org/10.31080/asms.2019.03.0357
21. Kovač Z. Pathophysiological body reactivity and interactions in comorbidities. Synergism versus antagonism of disease pathways and etiopathogenetic clusters in comorbidity conditions. Psychiatr Danub. 2021;33(4):414–26. PMID: 30946713
22. Pascart T, Latourte A, Flipo RM, Chales G, Coblentz-Baumann L, Cohen-Solal A, et al. 2020 recommendations from the French Society of Rheumatology for the management of gout: Urate-lowering therapy. Joint Bone Spine. 2020:87(5):395–404. https://doi.org/10.1016/j.jbspin.2020.05.002
23. Fitzgerald JD, Dalbeth N, Mikuls T, Brignardello-Petersen R, Guyatt G, Abeles AM, et al. 2020 American College of Rheumatology guideline for the management of gout. Arthritis Care Res (Hoboken). 2020;72(6):744–60. https://doi.org/10.1002/acr.24180
24. Громова МА, Цурко ВВ. Новые рекомендации Американской коллегии ревматологов по ведению больных подагрой (2020). Комментарии к некоторым позициям. РМЖ. Медицинское обозрение. 2021;5(2):89–95. https://doi.org/10.32364/2587-6821-2021-5-2-89-95
25. Драпкина ОМ, Мазуров ВИ, Мартынов АИ, Гайдукова ИЗ, Дупляков ДВ, Невзорова ВА и др. «В фокусе гиперурикемия». Резолюция Cовета экспертов. Кардиоваскулярная терапия и профилактика. 2023;22(4):3564. https://doi.org/10.15829/1728-8800-2023-3564
26. Yanai H, Adachi H, Hakoshima M, Katsuyama H. Molecular biological and clinical understanding of the pathophysiology and treatments of hyperuricemia and its association with metabolic syndrome, cardiovascular diseases and chronic kidney disease. Int J Mol Sci. 2021;22(17):9221. https://doi.org/10.3390/ijms22179221
27. Кобалава ЖД, Троицкая ЕА. Бессимптомная гиперурикемия: подходы к лечению в аспекте риска развития сердечно-сосудистых и почечных заболеваний. Кардиология. 2020;60(12):104–9. https://doi.org/10.18087/cardio.2020.12.n1158
28. Richette P, Clerson P, Perissin L, Flipo R-M, Bardin T. Revisiting comorbidities in gout: A cluster analysis. Ann Rheum Dis. 2015;74(1):142–7. https://doi.org/10.1136/annrheumdis-2013-203779
29. Lee JK, Kim JW, Kim YS, Koo BS. A case of severe gouty tophi-induced carpal tunnel syndrome: Operative finding and its outcome. Handchir Mikrochir Plast Chir. 2018;50(1):19–21. https://doi.org/10.1055/a-0576-7299
30. Lin GL, Lin HC, Lin HL, Keller J, Wang L-H, et al. Association between statin use and the risk of gout in patients with hyperlipidemia: A population-based cohort study. Front Pharmacol. 2023;14:1096999. https://doi.org/10.3389/fphar.2023.1096999
31. Akbari A, Razmi M, Rafiee M, Watts GF, Sahebkar A. The effect of statin therapy on serum uric acid levels: A systematic review and meta-analysis. Curr Med Chem. 2023;31(13):1726–39. https://doi.org/10.2174/0929867330666230207124516
32. Yip ASY, Leong S, Teo YH, Teo YN, Syn NLX, See RM, et al. Effect of sodium-glucose cotransporter-2 (SGLT2) inhibitors on serum urate levels in patients with and without diabetes: A systematic review and meta-regression of 43 randomized controlled trials. Ther Adv Chronic Dis. 2022;13:20406223221083509. https://doi.org/10.1177/20406223221083509
33. Stamp LK, Chapman PT, Barclay ML, Horne A, Frampton C, Tan P, et al. A randomised controlled trial of the efficacy and safety of allopurinol dose escalation to achieve target serum urate in people with gout. Ann Rheum Dis. 2017;76(9):1522–8. https://doi.org/10.1136/annrheumdis-2016-210872
34. Паневин ТС, Елисеев МС, Насонов ЕЛ. Уратснижающие препараты в лечении подагры: неизвестное об известном. Научно-практическая ревматология. 2021;59(6):727–37. https://doi.org/10.47360/1995-4484-2021-727-737
35. Гаранин АА, Новичкова НЛ, Лебедев ПА. Перспективы противовоспалительной и уратснижающей терапии подагры: вектор от прошлого к будущему. Научно-практическая ревматология. 2022;60(2):205–13. https://doi.org/10.47360/1995-4484-2022-205-213
36. Kell DB, Goodacre R. Metabolomics and systems pharmacology: Why and how to model the human metabolic network for drug discovery. Drug Discovery Today. 2014;19(2):171–82. https://doi.org/10.1016/j.drudis.2013.07.014
37. Ha J, Park H, Park J, Park SB. Recent advances in identifying protein targets in drug discovery. Cell Chemical Biology. 2021;28(3):394–423. https://doi.org/10.1016/j.chembiol.2020.12.001
38. Wishart DS. Emerging applications of metabolomics in drug discovery and precision medicine. Nat Rev Drug Discovery. 2016;15(7):473–84. https://doi.org/10.1038/nrd.2016.32
39. Kankanige D, Liyanage L, O’Connor MD. Application of transcriptomics for predicting protein interaction networks, drug targets and drug candidates. Front Med Technol. 2022;4:693148. https://doi.org/10.3389/fmedt.2022.693148
40. Pabon NA, Xia Y, Estabrooks SK, Ye Z, Herbrand AK, Süß E, et al. Predicting protein targets for drug-like compounds using transcriptomics. PLoS Comput Biol. 2018;14(12):e1006651. https://doi.org/10.1371/journal.pcbi.1006651
41. Таипов МА, Никифорова ЗН, Шевченко ВЕ. Протеомные исследования, направленные на поиск маркеров рака молочной железы (обзор литературы). Опухоли женской репродуктивной системы. 2015;11(2):8–18. EDN: TZZYKB
42. Manes NP, Nita-Lazar A. Application of targeted mass spectrometry in bottom-up proteomics for systems biology research. J Proteomics. 2018;189:75–90. https://doi.org/10.1016/j.jprot.2018.02.008
43. Kopec KK, Bozyczko-Coyne D, Williams M. Target identification and validation in drug discovery: the role of proteomics. Biochem Pharmacol. 2005;69(8):1133–9. https://doi.org/10.1016/j.bcp.2005.01.004
44. Moellering RE, Cravatt BF. How chemoproteomics can enable drug discovery and development. Chem Biol. 2012;19(1):11–22. https://doi.org/10.1016/j.chembiol.2012.01.001
45. Villanueva MT. Proteomics reveals small molecules’ secrets. Nat Rev Drug Discovery. 2023;22(3):181. https://doi.org/10.1038/d41573-023-00019-7
46. Wishart DS. Applications of metabolomics in drug discovery and development. Drugs R D. 2008;9(5):307–22. https://doi.org/10.2165/00126839-200809050-00002
47. Mastrangelo A, Armitage EG, García A, Barbas C. Metabolomics as a tool for drug discovery and personalised medicine. A review. Curr Top Med Chem. 2014;14(23):2627–36. https://doi.org/10.2174/1568026614666141215124956
48. Pun FW, Ozerov IV, Zhavoronkov A. AI-powered therapeutic target discovery. Trends Pharmacol Sci. 2023;44(9):561–72. https://doi.org/10.1016/j.tips.2023.06.010
49. Bai F, Li S, Li H. AI enhances drug discovery and development. Natl Sci Rev. 2023;11(3):nwad303. https://doi.org/10.1093/nsr/nwad303
50. Lv Q, Zhou F, Liu X, Zhi L. Artificial intelligence in small molecule drug discovery from 2018 to 2023: Does it really work? Bioorg Chem. 2023;141:106894. https://doi.org/10.1016/j.bioorg.2023.106894
51. Schaefer CF, Anthony K, Krupa S, Buchoff J, Day M, Hannay T, et al. PID: the Pathway Interaction Database. Nucleic Acids Research. 2009;37(1):674–9. https://doi.org/10.1093/nar/gkn653
52. Sadri A. Is target-based drug discovery efficient? Discovery and “off-target” mechanisms of all drugs. J Med Chem. 2023;66(18):12651–77. https://doi.org/10.1021/acs.jmedchem.2c01737
53. Tun K, Menghini M, D’Andrea L, Dhar P, Tanaka H, Giuliani A. Why so few drug targets: A mathematical explanation? Bentham Science. 2011;7(3):206–13. https://doi.org/10.2174/157340911796504297
54. Vincent F, Nueda A, Lee J, Schenone M, Prunotto M, Mercola M. Phenotypic drug discovery: Recent successes, lessons learned and new directions. Nat Rev Drug Discovery. 2022;21(12):899–914. https://doi.org/10.1038/s41573-022-00472-w
55. Balakin KV. Analysis of the structural transformations underlying the design of innovative next-in-class drugs. Russ J Gen Chem. 2023;93(Suppl 2):401–25. https://doi.org/10.1134/s107036322315001x
56. Britanova OV, Lupyr KR, Staroverov DB. Shagina IA, Aleksandrov AA, Ustyugov YY. Targeted depletion of TRBV9+ T cells as immunotherapy in a patient with ankylosing spondylitis. Nat Med. 2023;29(11):2731–6. https://doi.org/10.1038/s41591-023-02613-z
57. Makhoba XH, Viegas Jr C, Mosa RA, Viegas FPD, Pooe OJ. Potential impact of the multi-target drug approach in the treatment of some complex diseases. Drug Des Devel Ther. 2020;14:3235–49. https://doi.org/10.2147/DDDT.S257494
58. Ramsay RR, Popovic-Nikolic MR, Nikolic K, Uliassi E, Bolognesi ML. A perspective on multi-target drug discovery and design for complex diseases. Clin Transl Med. 2018;7(1):3. https://doi.org/10.1186/s40169-017-0181-2
59. Löscher W. Single-target versus multi-target drugs versus combinations of drugs with multiple targets: Preclinical and clinical evidence for the treatment or prevention of epilepsy. Front Pharmacol. 2021;12:730257. https://doi.org/10.3389/fphar.2021.730257
60. Sánchez-Tejeda JF, Sánchez-Ruiz JF, Salazar JR, Loza-Mejía MA. A definition of “multitargeticity”: Identifying potential multitarget and selective ligands through a vector analysis. Front Chem. 2020;8:176. https://doi.org/10.3389/fchem.2020.00176
61. Talevi A. Multi-target pharmacology: possibilities and limitations of the “skeleton key approach” from a medicinal chemist perspective. Front Pharmacol. 2015;6:205. https://doi.org/10.3389/fphar.2015.00205
62. Bizzarri M, ed. Approaching complex diseases. Human perspectives in health sciences and technology. Springer; 2020. https://doi.org/10.1007/978-3-030-32857-3
63. Lin HH, Zhang LL, Yan R, Lu JJ, Hu Y. Network analysis of drug–target interactions: A study on FDA-approved new molecular entities between 2000 to 2015. Sci Rep. 2017;7(1):12230. https://doi.org/10.1038/s41598-017-12061-8
64. Meerson A, Khatib S, Mahajna J. Natural products targeting cancer stem cells for augmenting cancer therapeutics. Int J Mol Sci. 2021 Dec 2;22(23):13044. https://doi.org/10.3390/ijms222313044
65. Lagunin A, Filimonov D, Poroikov V. Multi-targeted natural products evaluation based on biological activity prediction with PASS. Curr Pharm Des. 2010;16(15):1703–17. https://doi.org/10.2174/138161210791164063
66. Zheng C, Wang J, Liu J. Pei M, Huang C, Wang Y. System-level multi-target drug discovery from natural products with applications to cardiovascular diseases. Mol Divers. 2014;18(3):621–35. https://doi.org/10.1007/s11030-014-9521-y
67. Kurkin VA. Phenуlpropanoids from medicinal plants: Distribution, classification, structural analysis, and biological activity. Chemistry of Natural Compounds. 2003;39(2):123–53. https://doi.org/10.1023/A:1024876810579
68. Paul D, Sanap G, Shenoy S, Kalyane D, Kalia K, Tekade RK. Artificial intelligence in drug discovery and development. Drug Discov Today. 2021;26(1):80–93. https://doi.org/10.1016/j.drudis.2020.10.010
69. Persidis A. The benefits of drug repositioning. Drug Discov World. 2011;12(2):9–12.
70. Лебедев ПА, Волова ЛТ, Осина НК, Паранина ЕВ. Инфламмасома NLRP3 — новая универсальная мишень терапевтического воздействия у пациентов с бессимптомной гиперурикемией и подагрой. Рациональная фармакотерапия в кардиологии. 2024;20(3):331–9. https://doi.org/10.20996/1819-6446-2024-3018
71. Tong Y, Wang Z, Cai L, Lin L, Liu J, Cheng J. NLRP3 inflammasome and its central role in the cardiovascular diseases. Oxid Med Cell Longev. 2020;2020:4293206. https://doi.org/10.1155/2020/4293206
72. Strandberg TE, Kovanen PT. Coronary artery disease: ‘gout’ in the artery? Eur Heart J. 2021;42(28):2761–4. https://doi.org/10.1093/eurheartj/ehab276
73. Peng TC, Wang CC, Kao TW, et al. Relationship between hyperuricemia and lipid profiles in US adults. Biomed Res Int. 2015;2015:127596. https://doi.org/10.1155/2015/127596
74. Волова ЛТ, Пугачев ЕИ, Старикова ТВ, Лебедев ПА, Шафиева ИА, Кузнецов СИ и др. In vitro клеточная гиперурикемическая гемотест-система для определения цитокинового статуса пациентов с подагрическим артритом. Наука и инновации в медицине. 2024;9(1):14–21. https://doi.org/10.35693/SIM546016
75. Horvath P, Aulner N, Bickle M, Davies AM, Del Nery E, Ebner D, et al. Screening out irrelevant cell-based models of disease. Nat Rev Drug Discov. 2016;15(11):751–69. https://doi.org/10.1038/nrd.2016.175
Дополнительные файлы

|
1. Fig. 2. Aetiopathogenetic architecture of gout (fragment) | |
| Тема | ||
| Тип | Прочее | |
Скачать
(104KB)
|
Метаданные ▾ | |
Рецензия
Для цитирования:
Авдеева Е.В., Варина Н.Р., Рязанова Т.К., Куркин В.А., Исакова Н.В., Волова Л.Т., Полторецкий Д.А. Этиопатогенетическая архитектура разработки лекарственных средств (на примере этиопатогенеза подагры). Регуляторные исследования и экспертиза лекарственных средств. 2025;15(6):642-654. https://doi.org/10.30895/1991-2919-2025-724
For citation:
Avdeeva E.V., Varina N.R., Ryazanova T.K., Kurkin V.A., Isakova N.V., Volova L.T., Poltoretskii D.A. Aetiopathogenetic Architecture for Pharmaceutical Development (Using Gout as a Case Study). Regulatory Research and Medicine Evaluation. 2025;15(6):642-654. https://doi.org/10.30895/1991-2919-2025-724